Методы оптимизации ЛР1. Предобработка данных
 Скачать 0.51 Mb. Скачать 0.51 Mb.
|
|
| Студент гр. 9304 | | Ковалёв П.Д. |
| Преподаватель | | Жангиров Т.Р. |
Санкт-Петербург
2022
Цель работы.
Ознакомиться с методами предобработки данных из библиотеки Scikit Learn.
Задание.
Загрузка данных.
Загрузить датасет по ссылке: https://www.kaggle.com/datasets/andrewmvd/heart-failure-clinical-data. Данные представлены в виде csv таблицы.
Создать Python скрипт. Загрузить датасет в датафрейм, и исключить бинарные признаки и признак времени.
Построить гистограммы признаков.
На основании гистограмм определите диапазоны значений для каждого из признаков, а также возле какого значения лежит наибольшее количество наблюдений.
Так как библиотека Sklearn работает с NumPy массива, то преобразуйте датафрейм к двумерному массиву NumPy, где строка соответствует наблюдению, а столбец признаку.
Стандартизация данных
Подключите модуль Sklearn. Настройте стандартизацию на основе первых 150 наблюдений используя StandardScaler.
Стандартизируйте все данные
Постройте гистограммы стандартизированных данных
Сравните данные до и после стандартизации. Опишите, что изменилось и почему.
Рассчитайте мат. ожидание и СКО до и после стандартизации. На основании этих значений выведите для каждого признака формулы по которым они стандартизировались.
Сравните значений из формул с полями mean_ и var_ объекта scaler
Проведите настройку стандартизации на всех данных и сравните с результатами настройки на основании 150 наблюдений
Приведение к диапазону
Приведите данные к диапазону используя MinMaxScaler
Постройте гистограммы для признаков и сравните с исходными данными
Через параметры MinMaxScaler определите минимальное и максимальное значение в данных для каждого признака
Аналогично трансформируйте данные используя MaxAbsScaler и RobustScaler. Постройте гистограммы. Определите к какому диапазону приводятся данные.
Напишите функцию, которая приводит все данные к диапазону [-5 10]
Нелинейные преобразования
Приведите данные к равномерному распределению используя QuantileTransformer
Постройте гистограммы и сравните с исходными данными
Определите, как и на что влияет значение параметра n_quantiles
Приведите данные к нормальному распределению передав в QuantileTransformer параметр output_distribution=‘normal’
Постройте гистограммы и сравните с исходными данными
Самостоятельно приведите данные к нормальному распределению используя PowerTransformer
Дискретизация признаков
Проведите дискретизацию признаков, используя KBinsDiscretizer, на следующее количество диапазонов:
age – 3
creatinine_phosphokinase – 4
ejection_fraction – 3
platelets – 10
serum_creatinine – 2
serum_sodium – 4
Постройте гистограммы. Объясните полученные результаты.
Через параметр bin_edges_ выведите диапазоны каждого интервала для каждого признака.
Выполнение работы.
Загрузка данных.
Был загружен датасет.
Далее был создан Python скрипт, после чего датасет был загружен в датафрейм с последующим исключением бинарных признаков и признака времени. Результат можно видеть на рисунке 1.
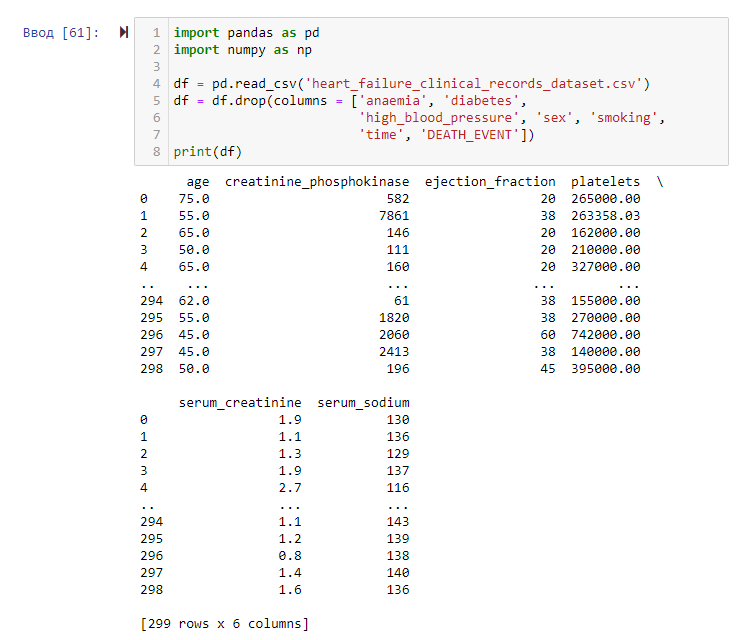
Рисунок 1 – Загрузка датасета и удаление признаков
Далее были построены гистограммы признаков, которые можно видеть на рисунке 2.
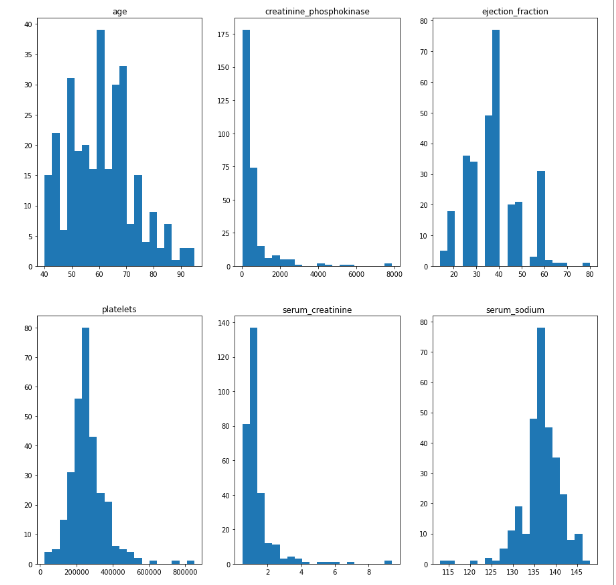
Рисунок 2 – Гистограммы признаков
На основании гистограмм были определены диапазоны значений дли каждого из признаков, а также возле какого значения лежит наибольшее количество наблюдений. Имеем:
Для age: диапазон значений от 40 до 95, наибольшее количество наблюдений лежит около значения 60.
Для creatinine_phosphokinase: диапазон значений от 0 до 8000, наибольшее количество наблюдений лежит около значения 0.
Для ejection_fraction: диапазон значений от 15 до 80, наибольшее количество наблюдений лежит около значения 37.
Для platelets: диапазон значений от 0 до 900000, наибольшее количество наблюдений лежит около значения 250000.
Для serum_creatinine: диапазон значений от 0 до 9, наибольшее количество наблюдений лежит около значения 1.
Для serum_sodium: диапазон значений от 0 до 150, наибольшее количество наблюдений лежит около значения 137.
Датафрейм был преобразован к двумерному массиву NumPy. Это можно видеть на рисунке 3.
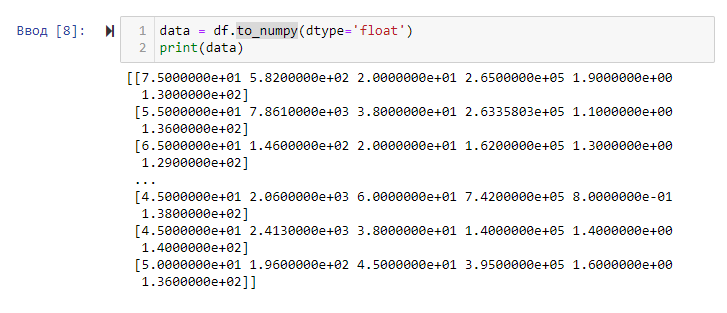
Рисунок 3 – Преобразование датафрейма в массив NumPy
Стандартизация данных.
Был подключен модуль Sklearn и настроена стандартизация на основе первых 150 наблюдений.
Все данные были стандартизированы.
Были построены гистограммы стандартизированных данных. Их можно видеть на рисунке 4.
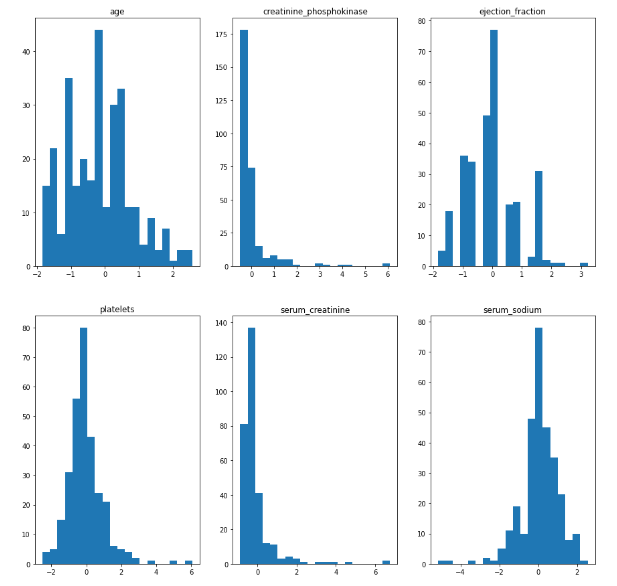
Рисунок 4 – Гистограммы стандартизированных данных
Наибольшее количество наблюдений лежит в пределах 0, при этом, диапазоны значений значительно уменьшились, чаще всего встречается диапазон от -2 до 2. Стандартизация приводит все исходные значения набора данных, независимо от их начальных распределений и единиц измерения, к набору значений из распределения с нулевым средним и стандартным отклонением, равным 1. Стандартизированное значение вычисляется по формуле:
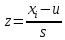 ,
,где
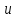 – мат. ожидание, а
– мат. ожидание, а 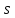 – среднеквадратичное отклонение.
– среднеквадратичное отклонение.Были рассчитаны мат.ожидания и СКО до и после стандартизации. Результаты представлены на рисунке 5.
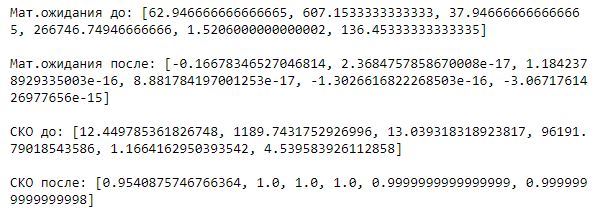
Рисунок 5 – Мат.ожидания и СКО до и после стандартизации
На рисунке 6 представлены значения полей mean_ и var_ объекта scaler.

Рисунок 6 – Значения полей mean_ и var_
Была проведена настройка стандартизации на всех данных. Результат представлен на рисунке 7.
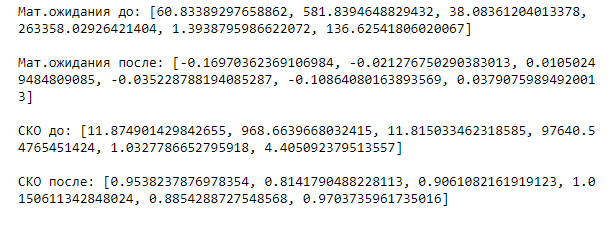
Рисунок 7 – Стандартизация всех данных
Как можно видеть, в случае стандартизации всех данных, значения мат.ожиданий и СКО несколько отклонились от значений, полученных при стандартизации первых 150 значений.
Приведение к диапазону.
Данные были приведены к диапазону при помощи MinMaxScaler.
Были построены гистограммы. Их можно видеть на рисунке 8.
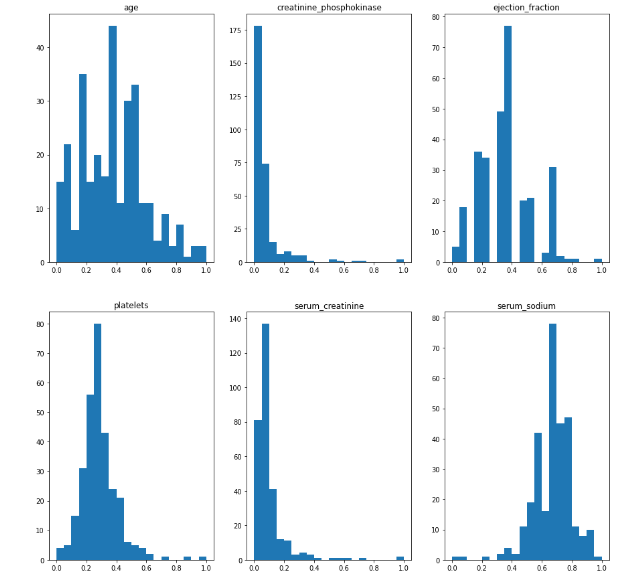
Рисунок 8 - Гистограммы
По сравнению с исходными данными все диапазоны находятся в пределах от 0 до 1.
Через параметры MinMaxScaler были определены минимальное и максимальное значения в данных для каждого признака. Это можно видеть на рисунке 9.
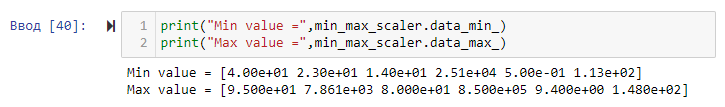
Рисунок 9 – Минимальное и максимальное значения в данных
На рисунках 10 и 11 представлены гистограммы данных, стандартизированных при помощи MaxAbsScaler и RobustScaler соответственно.
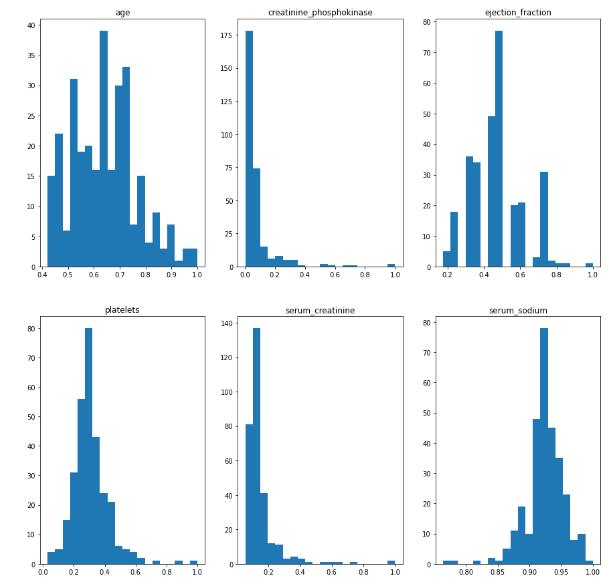
Рисунок 10 – Гистограмма с использованием MaxAbsScaler
Масштабирование привело данные к диапазону от 0 до 1. MaxAbsScaler масштабирует данные по максимальному абсолютному значению.
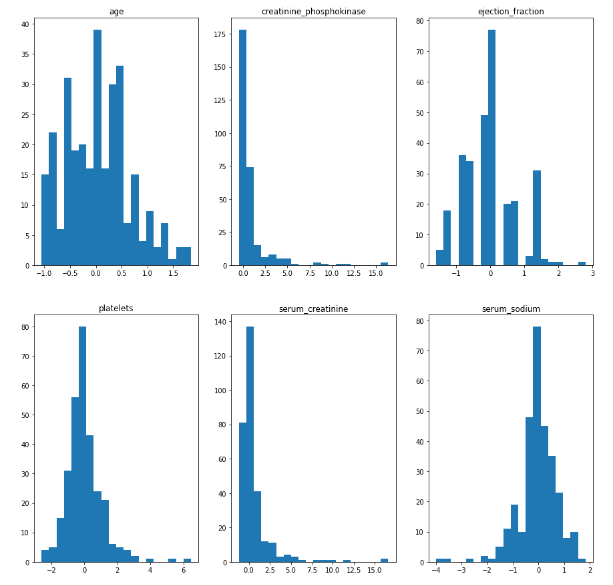
Рисунок 11 – Гистограмма с использованием RobustScaler
Robust Scaler удаляет медиану и масштабирует данные в соответствии с диапазоном квантилей.
На рисунке 12 представлен код функции и её вызов для отрисовки гистограмм, а на рисунке 13 представлены сами гистограммы.
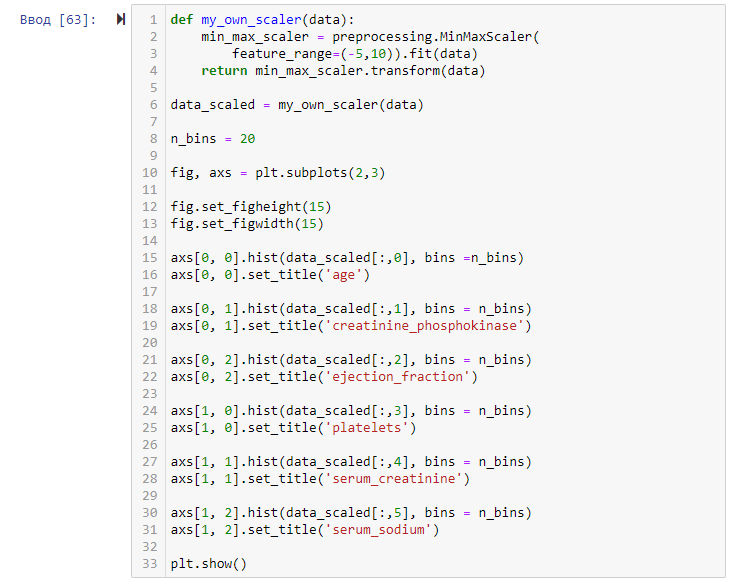
Рисунок 12 – Код функции и её вызова
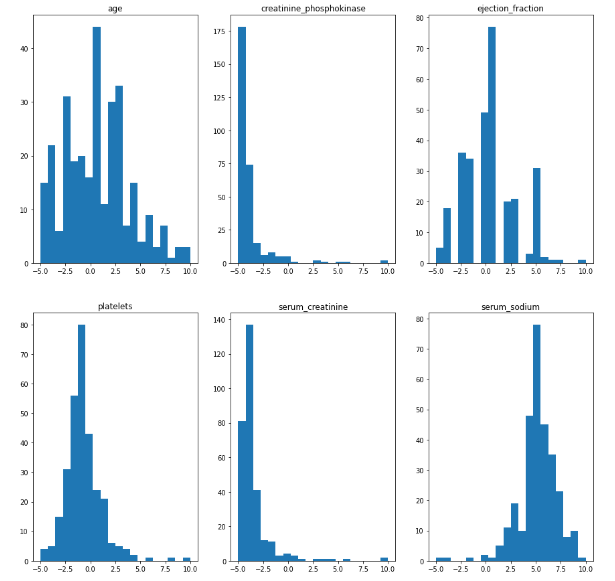
Рисунок 13 – Гистограммы
Нелинейные преобразования.
Данные были приведены к равномерному распределению при помощи QuantileTransformer.
Были построены гистограммы данных. Это можно видеть на рисунке 14.
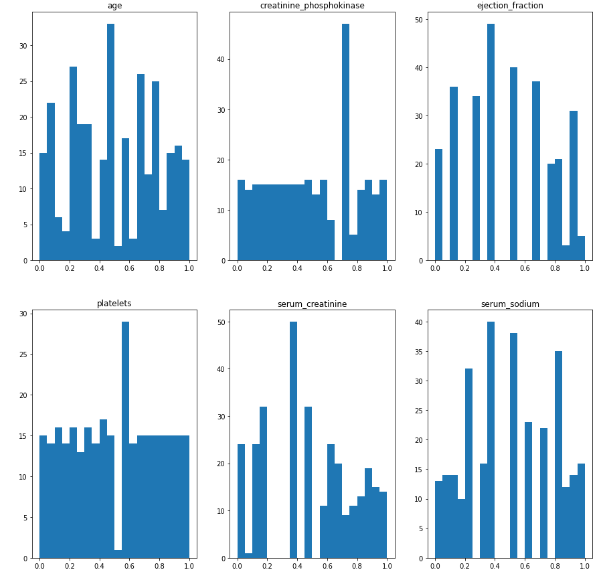
Рисунок 14 – Гистограммы
Данные лежат в диапазон от 0 до 1 и имеют равномерное распределение.
Параметр n_quantiles влияет на количество вычисляемых квантилей.
Данные были приведены к нормальному распределению.
На рисунке 15 представлены гистограммы с нормальным распределением.
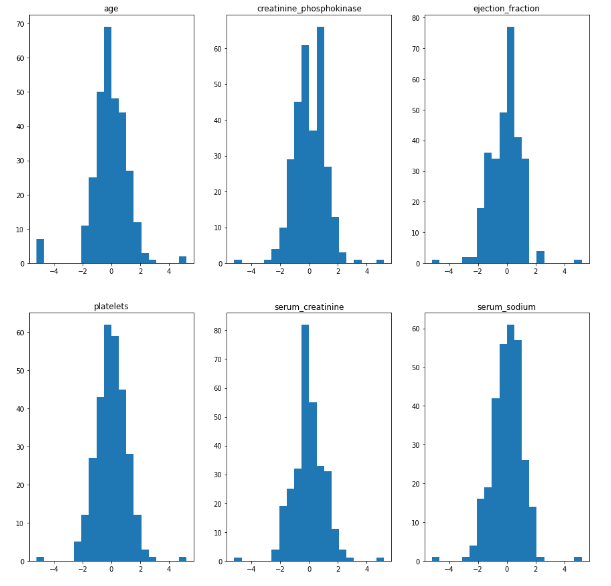
Рисунок 15 – Гистограммы
По гистограммам видно, что данные имеют нормальное распределение.
Данные были приведены к нормальному (Гауссовому) распределению при помощи PowerTransformer(). Результаты представлены на рисунке 16.
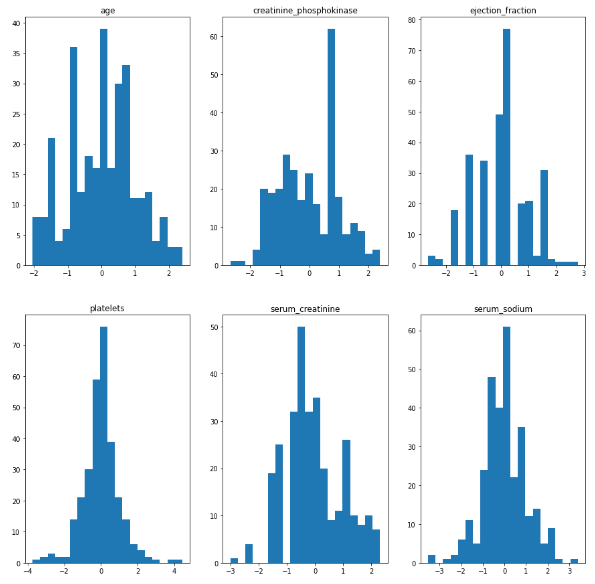
Рисунок 16 – Гистограммы
Дискретизация признаков.
Была проведена дискретизация признаков при помощи KBinsDiscretizer.
На рисунке 17 представлены гистограммы дискретизации признаков.
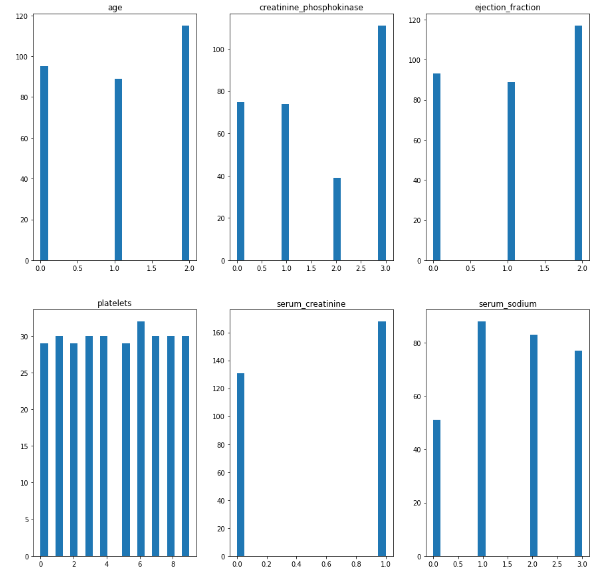
Рисунок 17 – Гистограммы
По гистограммам видно, что данные были разделены на интервалы.
На рисунке 18 представлены диапазоны каждого интервала для каждого признака.
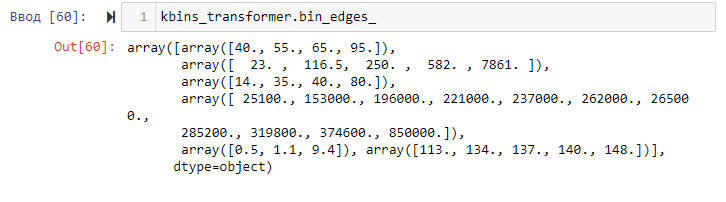
Рисунок 18 – Диапазоны интервалов
Выводы.
Были изучены предоставленные библиотекой Scikit-learn средства для стандартизации данных, средства нелинейных преобразований и метод дискретизации данных, которые в процессе были опробованны на наборе данных.
