Методы аналитической биохимии. 1. Правила проведения работ в лаборатории при проведении биохимического анализа с использованием лабораторных животных
 Скачать 1.66 Mb. Скачать 1.66 Mb.
|
|
32. Рестрикционный анализ. Полиморфизм длин рестрикционных фрагментов (ПДРФ) — это способ исследования геномной ДНК, путем разрезания ДНК с помощью эндонуклеаз рестрикции и дальнейшего анализа размеров образующихся фрагментов (рестриктов) путем гель-электрофореза. При использовании данного исследования получаются различные результаты от различных образцов, и при помощи анализа полиморфизма длин фрагментов рестрикции можно идентифицировать некоторые различия в последовательности нуклеотидов ДНК, в случае, когда они располагаются в сайте рестрикции. 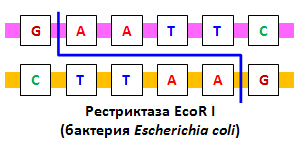 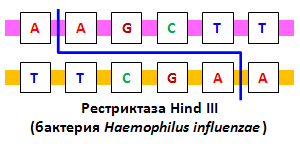 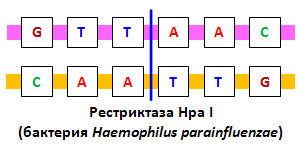 Наличие сайтов рестрикции в геномной ДНК и их взаимное расположение однозначно определяются последовательностью нуклеотидов исследуемой ДНК, поскольку сам сайт рестрикции — не что иное, как строго определенная последовательность нуклеотидов ДНК, узнаваемая и расщепляемая рестриктазами. Следовательно, любая мутация, изменяющая последовательность нуклеотидов сайта рестрикции, уничтожает этот сайт. Полное расщепление анализируемой геномной ДНК отдельными рестриктазами приводит к образованию определенного набора фрагментов ДНК, число и размеры которых соответствуют расположению сайтов рестрикции. Мутационная изменчивость в сайтах рестрикции может быть легко обнаружена по изменению длины рестрикционных фрагментов ДНК. При наличии мутации в одном из сайтов рестрикции этот сайт остается нерасщепленным после завершения рестрикции, что приводит к слиянию соседних рестрикционных фрагментов ДНК, разделяемых мутантным сайтом, и образованию фрагмента ДНК большего размера. В результате длина рестрикционных фрагментов ДНК, содержащих мутантные сайты, становится полиморфной, что выявляется при сравнении ДНК из разных источников методом ПДРФ. Полиморфизм ДНК в нашем случае это наличие вариабельности в последовательности нуклеотидов в одном и том же гене (выполняющем те же функции) у близкородственных видов. Полиморфизм вызывается несколькими причинами: точечными мутациями в виде единичных нуклеотидных замен, ошибками при репликации ДНК в виде инсерций или делеций протяжённостью от одного до сотен или тысяч нуклеотидов, крупными делециями, вставками, транслокациями, транспозициями мобильных генетических элементов и т. п. Все изменения в первичной структуре ДНК ведут к изменениям в длине фрагментов, образующихся под воздействием рестриктаз. Эндонуклеазы рестрикции Эндонуклеазы рестрикции – своеобразные молекулярные ножницы – это ферменты, которые разрезают фосфодиэфирную связь между соседними нуклеотидами в молекуле ДНК. Разные типы эндонуклеаз рестрикции различаются между собой по особенностям узнавания и разрезания молекул ДНК. Эндонуклеазы рестрикции II типа – ферменты, которые распознают в молекулах ДНК короткие (4-14 пар нуклеотидов) зеркально симметричные участки (так называемые палиндромы) и разрезают обе цепи ДНК посередине этих участков либо со смещением. Эти участки называются сайтами рестрикции. Каждая эндонуклеаза рестрикции II типа характеризуется своим особенным сайтом рестрикции и уникальным способом разрезания. В настоящее время для решения генно-инженерных задач используется несколько сотен эндонуклеаз рестрикции II типа. Название рестриктазы образуется путем сочетания первой буквы рода бактерии и двух первых букв вида бактерии, из которых они были выделены. За основу берется название бактерии на латинском языке. Например, фермент EcoRI назван так, потому что выделен из бактерии Escherichia coli штамма RY13 первым по счету (I). Эндонуклеазы рестрикции разрезают молекулу ДНК либо по центру сайта рестрикции, либо со смещением. В первом случае, в результате разрезания образуются двухцепочечные («тупые») концы, а во втором – фрагменты, которые содержат активные одноцепочечные участки ДНК. Именно эти одноцепочечные участки будут стремиться при первой же возможности снова объединиться с комплементарными участками, из-за чего их и называют «липкими» концами. 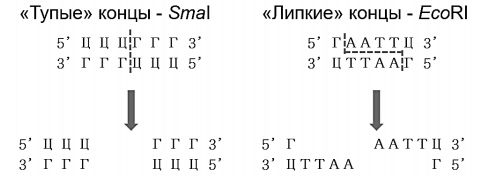 Следует отметить, что для успешного разрезания молекул ДНК эндонуклеазами рестрикции II типа важно именно наличие подходящего сайта рестрикции, но не происхождение самой молекулы ДНК. Таким образом, ферменты одинаково хорошо разрежут ДНК герани, стрептококка, жирафа, вируса гриппа, сыроежки и человека. Способность эндонуклеаз рестрикции II типа разрезать любую по происхождению ДНК, а также описанные выше свойства «липких» концов ученые активно используют для создания рекомбинантных ДНК. 33. ПЦР в режиме реального времени. ПЦР в реальном времени (Real-time PCR)–метод позволяет регистрировать накопление ДНК ПЦР в реальном времени с использованием флуоресценции и количественно оценить ДНК в образце. Для этой цели применяют флуоресцентные метки (флуорофоры) двух видов: 1) Интеркалирующие флуорофоры – соединения, которые встраиваются в любые двуцепочечные молекулы нуклеиновой кислоты и повышают интенсивность флуоресценции. Примерами таких красителей может служить SYBR Green или SYBR Gold. Интеркалирующие красители широко используются в научных исследованиях, однако обладают существенным ограничением, поскольку регистрируют все двуцепочечные ДНК, включая неспецифичные продукты реакции. 2) Флуорофоры для меченья олигонуклеотидов – это соединения, интенсивность флуоресценции которых не зависит от связывания с ДНК и регулируется так называемыми гасителями (темновыми или флуоресцирующими). Олигонуклеотиды меченные парой флуорофор-гаситель, используют в качестве праймеров или зондов (пробы) для гибридизации со специфическим участком молекулы ДНК. К настоящему времени разработано множество вариантов структуры меченных парой флуорофор – гаситель олигонуклеотидов. Ниже приведены наиболее часто используемые зонды. Линейные разрушаемые зонды (TaqMan) – представляют собой олигонуклеотид (25-30 н.о.), в котором 5’-концевой нуклеотид содержит флуорофор, а 3’-концевой нуклеотид-гаситель. В растворе и при отжиге на ДНК такой зонд не флуоресцирует, за сет гасителя. Во время элонгации ДНК полимераза, обладающая 5’-3’-экзонуклеазной активностью, гидролизует зонд на нуклеотиды. В результате этого флуорофор и гаситель попадают в раствор, где вероятность нахождения этих веществ рядом будет небольшой, и флуоресценция восстановится. Накопление флуоресценции детектируется прибором при каждом цикле ПЦР. В одной реакции можно использовать несколько зондов, у которых флуорофоры имеют разные спектры испускания. «Молекулярные маячки» (beacons) – зонд представляет собой небольшую одноцепочечную молекулу ДНК, которая в свободном состоянии способна образовывать пространственную структуру – шпильку. На одном конце олигонуклеотида располагают флуорофор, а на втором – гаситель. Последовательность зонда комплементарна ДНК-мишени, которую нужно детектировать. В такой структуре молекулы зонда, находясь в растворе, не флуоресцируют. При отжиге олигонуклеотида на молекуле ДНК мишени, происходит пространственное разнесение флуорофора от гасителя и восстановление флуоресценции. Примыкающие пробы (Lightcyaler) – в этом варианте используют два зонда, которые связывают ДНК-мишень на небольшом расстоянии друг от друга. 5’-конец одного зонда и 3’-конец второго содержат флуорофор-донор и флуорофор-акцептор, соответственно. При их близком расположении флуорофор-донор поглощает свет определенной длины волны и переносит энергию на флуорофор-акцептор, по флуоресценции которого детектируют продукты амплификации. Применение примыкающих проб также позволяет использовать несколько флуорофоров, с разными спектрами испускания и детектировать в одной пробирке сигнал от разных молекул ДНК. Анализ данных ПЦР в реальном времени позволяет судить как о присутствии или отсутствии искомой НК, так и оценить ее изначальное количество, за счет регистрации накопления продуктов амплификации в течение всей реакции. Количество исследуемой НК выражают в абсолютных или относительных значениях. Определение абсолютных значений применяют для расчета количества копий НК в пробирке. Для этого необходим внешний контроль с известной концентрацией НК-мишени. Относительные количества определяют для ответа на вопрос: во сколько раз концентрация НК в одном образце больше (или меньше) чем в другом? Для коррекции массы образца, числа клеток, сохранности и количества общей НК проводят нормировку (выравнивание) результатов. Нормировку проводят по числу клеток, по суммарной ДНК, РНК, рибосомальной РНК и др. В исследованиях по определению уровня представленности транскриптов наибольшее распространение получило использование так называемых генов «домашнего хозяйства» (housekeeping genes). Они экспрессируются на постоянном уровне и слабо реагируют на внешние воздействия, поскольку необходимы для поддержания важнейших жизненных функций клетки. При выборе гена домашнего хозяйства, следует учитывать, что уровень их экспрессии может меняться в разных типах клеток. Расчеты результатов ПЦР в реальном времени достаточно сложны и проводятся в автоматическом режиме с использованием компьютерных программ. Одной из программ для расчета относительного количества ДНК-мишени, находящейся в свободном доступе в сети Интернет, является REST2009. Расчеты основаны на связи интенсивности флуоресцентного сигнала с концентрацией ДНК в пробирке. Сравнивают число циклов амплификации, необходимых для нарастания сигнала флуоресценции в образцах до одной и той же величины. Теоретически концентрация ампликонов при многократном повторении цикла должна возрастать экспоненциально и описывается формулой (1): Сn = С0 x 2n (1) где С0 – исходное количество ДНК-мишени, Сn – концентрация ампликонов после n циклов На практике, синтез ампликонов постепенно замедляется по мере истощения компонентов реакционной смеси. Накопление ДНК в ходе ПЦР описывают кривой (рис. 1) и делят на четыре стадии: линейная фоновая фаза, стадия раннего экспоненциального роста количества ампликона, стадия линейного логарифмического роста, стадия выхода на плато. На стадии раннего экспоненциального роста эффективность амплификации максимальна в сравнении с другими стадиями. Накопление ДНК в ходе ПЦР описывается формулой (2): Сn = С0 х (1 + E)n (2) где Сn – количество продуктов реакции на цикле n; C0 – изначальное количество исследуемой ДНК на первом цикле; Е – эффективность амплификации. Значение Е находится в пределах от 0 до 1, что соответствует удвоению исходного количества ДНК за цикл, а при Е = 0 образование продукта не происходит. Для сравнения данных между образцами используют один из двух методов. В первом, для расчета величины n используют значение порогового цикла (Ct – от англ. threshold cycle) – точки пересечения кривой накопления флуоресценции и одинаковой для всех образцов пороговой линии. Значение Ct определяется автоматически программным обеспечением к прибору или задается вручную в пределах стадии раннего экспоненциального роста накопления флуоресценции. Во втором, используют метод прямого сравнения графиков. Для определения величины n на кривой находят точку, положение которой отражает форму кривой (Ср – от англ. crossing point). Обычно рассчитывают максимумы первой и второй производных графиков накопления флуоресценции. Значение эффективности амплификации подбирается эмпирически путем последовательных разбавлений образца. 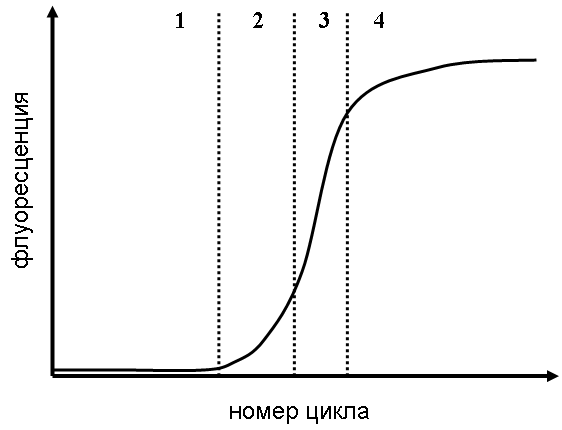 Рис. 1. Зависимость флуоресценции от номера цикла: 1 – Линейная фоновая фаза. 2 – Ранний экспоненциальный рост. 3 – Линейный логарифмический рост. 4 – Плато ПЦР в реальном времени обладает рядом преимуществ, за счет которых с его помощью, например, выявляют и идентифицируют полиморфные локусы HLA, проводят мониторинг посттрансплантационных взаимодействий, генотипируют (выявляют аллели), проводят количественный микросателлитный анализ и пренатальную диагностику генетических заболеваний и т.д. 34. Секвенирование ДНК. Полногеномное секвенирование. Секвенирование нуклеиновых кислот (ДНК и РНК) – метод, позволяющий получить описание первичной структуры линейной макромолекулы в виде последовательности мономеров (нуклеотидов). Определение нуклеотидной последовательности ДНК стало возможным во второй половине 1970-х годов благодаря стремительному развитию методов молекулярной биологии (использование эндонуклеаз рестрикции, плазмидных векторов, молекулярного клонирования и электрофоретического разделения нуклеиновых кислот и т.д.). Первыми методами секвенирования были метод, основанный на специфической химической деградации фрагмента ДНК, разработанный А. Максамом и В. Гилбертом, и дидезоксинуклеотидный метод, предложенный Ф. Сэнгером. Метод Сэнгера, в модифицированном и более технологичном виде, получил наибольшее распространение. С развитием самой молекулярной биологии и смежных областей (химия, физика и информатика) были разработаны методы секвенирования нового поколения (next-generation sequencing – NGS), отличающиеся более высокой производительностью. Наиболее производительные методы, обеспечивают чтение миллиардов нуклеотидов в день. Однако, несмотря на наличие технологий с высокой пропускной способностью, дидезоксинуклеотидный метод до сих пор остается «золотым стандартом» определения нуклеотидной последовательности ДНК и широко используется в научно-исследовательских работах. Секвенирование ДНК дидезоксинуклеотидным методом по Сэнгеру В основе метода лежит ферментативный синтез комплементарной цепи ДНК с использованием так называемых «терминаторов» или терминирующих дидезоксинуклеотидов – 2'- и 3'-дидезоксинуклеозидтрифосфатов (ддНТФ: ддATФ, ддTTФ, ддЦTФ и ддГTФ), которые включаясь в растущую цепь ДНК, препятствуют (терминируют) присоединение следующего нуклеотида из-за отсутствия 3'-ОН группы. По теории вероятности, при определенном отношении концентраций дНTФ/ддНTФ и избытке молекул секвенируемой ДНК, синтез строящейся цепи может остановиться на каждом нуклеотиде исследуемого фрагмента ДНК. В итоге синтезируется набор фрагментов ДНК различной длины, 3'-конец которых заканчивается одним из терминаторов. Ф.Сэнгером было предложено проводить четыре реакции для каждого из ддНТФ и анализировать результаты реакции в полиакриламидном геле на соседних дорожках. Нуклеотидная последовательность считывалась по расположению полос на геле. В настоящее время дидезоксинуклеотидный метод секвенирования ДНК по Сэнгеру, известный так же как метод обрыва растущей цепи, претерпел многочисленные модификации, основанные на современных достижениях (рис. 1). Ферментативный синтез комплементарной цепи ДНК проводится в ходе ПЦР, в которой наряду с дНТФ используют ддНТФ. Каждый из ддНТФ помечен отдельным флуоресцентным красителем (флуорофором), что позволяет проводить реакцию в одной пробирке. Результатом такой ПЦР является синтез пула одноцепочных фрагментов ДНК разного размера и заканчивающихся одним из флуоресцентно-меченных ддНТФ. Полученные фрагменты ДНК очищают от компонентов реакционной смеси, денатурируют и разделяют по длине в линейном полиакриламидном геле методом электрофореза. Используют высоковольтный капиллярный электрофорез, позволяющий разделять фрагменты ДНК, отличающиеся всего на один нуклеотид. 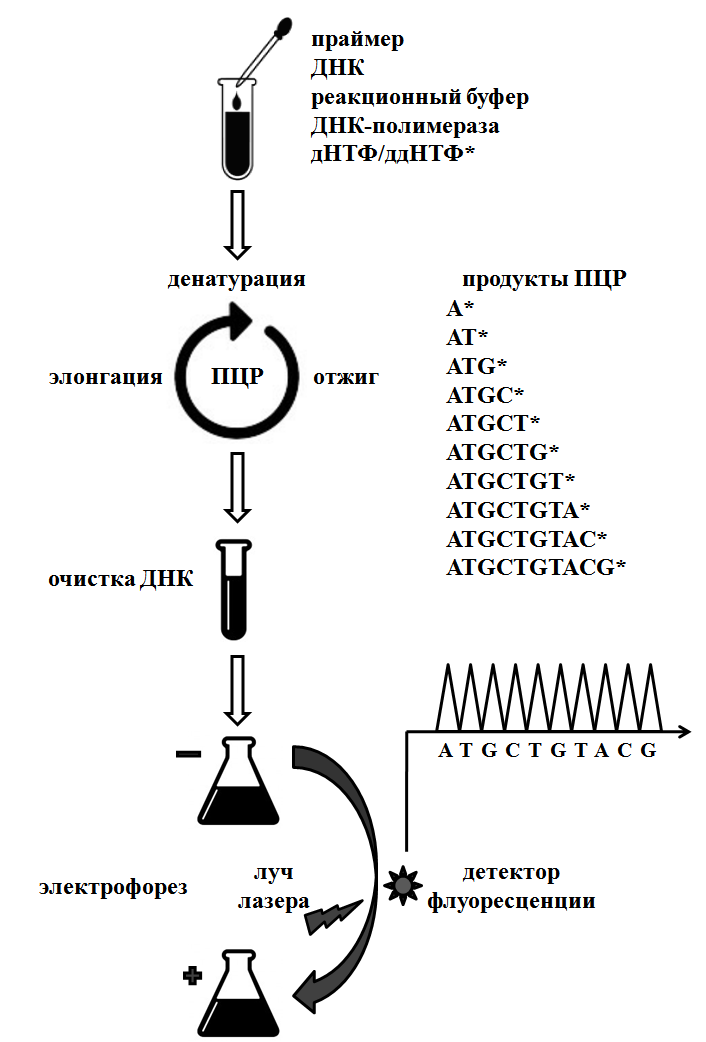 Рисунок 1 – Схема проведения секвенирования ДНК по Сэнгеру Электрофоретическое разделение фрагментов ДНК проводят в автоматических капиллярных секвенаторах («GE – MegaBACE», «Beckman Coulter – CEQ», «Applied Biosystems»). При прохождении меченного флуоресцентным красителем фрагмента через зону сканирования лазер, генерирующий непрерывное излучение, возбуждает флуорофор, следующая за этим эмиссия (излучение) флуоресценции улавливается детектирующим устройством. Полученные данные преобразуется с помощью программного обеспечения в текстовую информацию (нуклеотидную последовательность). Определение нуклеотидной последовательности ДНК по Сэнгеру является самым точным методом секвенирования, но обладает небольшой производительностью. Например, проект «Геном человека», занял приблизительно 13 лет и стоил порядка 3 миллиардов долларов. Однако, разработанные при реализации проекта технические решения, привели к стремительному развитию технологий секвенирования следующего поколения. Например, способ подготовки ДНК, названный методом «дробовика» («shotgun-sequencing»), послужил основой для создания массивного параллельного секвенирования, используемого в следующих поколениях секвенирования (NGS). Следующие поколения методов секвенирования Технологии NGS обладают высокой производительностью, скоростью, остаточно высокой точностью и позволяют решать разнообразные задачи (полногеномное секвенирование, секвенирование геномов и транскриптомов de novо, ресеквенирование с поиском заданной мутации, метагеномные исследования и т.д.). Большое значение для NGS играет биоинформатика, т.к. обработка полученных данных и сборка нуклеотидных последовательностей требует высокопроизводительного программного обеспечения. Массивное параллельное секвенирование является новым этапом в совершенствовании технологий определения нуклеотидных последовательностей. Часто их относят к методам второго поколения NGS. Принципиальное отличие технологий массивного параллельного секвенирования состоит в возможности одновременного определения первичной структуры пула нуклеиновых кислот, выделенных из различных образцов. Для дифференцировки исследуемых проб используют наборы штрих-кодов или индексов, представляющих собой олигонуклеотиды известной последовательности. Массивное параллельное секвенирование Данное исследование представлено четырьмя основными технологиями: Технология 454 (Roche, Швейцария), известная так же как пиросеквенирование. Принцип технологии основан на детекции хемилюминесцентного сигнала, образующегося в результате высвобождения пирофосфата при встраивании ДНК-полимеразой соответствующего нуклеотида в процессе синтеза комплементарной цепи ДНК (http://www.454.com/ ). Секвенирование на молекулярных кластерах (Illumina, США). Технология основана на детекции флуоресцентного сигнала, полученного при встраивании ДНК-полимеразой в растущую цепь ДНК одного из четырех типов дНТФ, отмеченных соответствующим флуорофором (http://www.illumina.com/ ). Полупроводниковая технология секвенирования (Ion Torrent/ Life Technologies, США). Принцип технологии основан на детекции изменения рН при выделении одного протона водорода в результате встраивания дНТФ ДНК-полимеразой в реальном времени (http://www.iontorrent.com/ ). Циклическое лигазное секвенирование (SOLiD/ Life Technologies, США). Принцип метода основан на сшивании (лигировании) ДНК-лигазой комплементарных одноцепочечной матрице ДНК флуоресцентных зондов (восьмичленных олигонуклеотидов) друг с другом и высвобождении при этом флуоресцентной метки, которая детектируется устройством (http://www.lifetechnologies.com/ ). Секвенирование третьего поколения Next-Next Generation Sequencing (NNGS) – основано на определении первичной структуры единичных молекул и характеризуется отсутствием стадии амплификации фрагментов ДНК. Представлено тремя технологиями: Секвенирование одной молекулы (true Single Molecule Sequencing, tSMS; Helicos BioSciences, США). Принцип метода основан на построении комплементарной фрагменту ДНК цепи при добавлении меченых нуклеотидов, при этом в местах связывания детектируется свечение, после чего метки от присоединенных нуклеотидов удаляют и вводят следующий тип меченых нуклеотидов. В настоящее время компания Helicos BioSciences прекратила свое существование, и данная технология не поддерживается. Секвенирование единичных молекул в реальном времени (Single Molecule RealTime, SMRT; Pacific BioSciences, США). Принцип метода основан на достройке в режиме реального времени комплементарной однонитевой матрице цепи ДНК-полимеразой, закрепленной на дне специальных ячеек, при этом сигнал от каждого присоединившегося нуклеотида, помеченного определенной светящейся меткой, фиксируется прибором (http://www.pacificbiosciences.com/ ). Секвенирование через нанопоры (Oxford Nanopore Technologies, Англия). Принцип метода основан на протягивании через нанопору отрицательно заряженного одноцепочечного фрагмента ДНК, при этом регистрируется изменение электропроводности нанопоры с помощью электродов по мере прохождения через нее нуклеотидов, каждому из которых (в силу физического различия нуклеотидных оснований) соответствует определенное изменение электропроводности (https://nanoporetech.com/ ). Существуют также другие варианты реализации секвенирования ДНК через нанопоры, но в настоящее время научно-исследовательские разработки нанопорных секвенаторов находятся на опытно-конструкторской стадии. Таким образом, современные методы секвенирования продолжают активно развиваться и совершенствоваться для выполнения разноплановых задач, связанных с определением нуклеотидной последовательности ДНК, а так же с целью увеличения производительности, скорости и точности секвенирования. |
